近期,浙江大学农业与生物技术学院在Genomics Communications 发表了题为Unlocking rice's genetic potential: big data-driven insights from population genomics 的综述论文。该文总结了水稻群体基因组学的研究进展,重点介绍了大规模重测序数据和泛基因组、基因组多样性对驯化和适应机制的解码以及将这些见解转化为精确育种策略。

随着测序技术的不断进步,水稻群体基因组学的研究得到了有力推动。第三代测序(TGS)技术以其长读长优势,突破了传统短读长测序的限制,成功实现了“日本晴”品种的无间隙基因组组装。2022年,水稻泛基因组研究整合了111个高质量基因组,涵盖了879 Mb的序列和19,000个基因。到了2025年,利用145份材料,研究人员绘制了迄今为止分辨率最高的野生稻与栽培稻泛基因组图谱。与此同时,多个水稻多组学数据库,如RiceVarMap、RiceSuperPIRdb和RPAN等,也在不断发展完善。此外,基于人工智能(AI)的平台,例如CropGS-Hub和Smart Breeding Platform等,通过机器学习(ML)和深度学习(DL)技术优化育种决策,实现了从数据存储到智能分析的完整闭环。
群体基因组学为深入解析水稻农艺性状提供了丰富的数据支持。多组学关联分析策略成功鉴定出许多关键基因,例如OsAmy3D通过调节淀粉消化速率影响血糖指数,OsWRKY19和OsNAC021则调控香气合成。结构变异(SVs)的功能验证也取得了重要进展,例如LTR插入导致Os06g13470上游变异,从而引发叶片早衰;4-kb插入破坏了LOC_Os06g04820的功能,进而降低了粒长。这些发现直接推动了预测育种的发展:基于卷积神经网络(CNN,如DeepGS、DNNGP)和Transformer(如GPformer、Cropformer)架构的模型显著提高了性状预测的精度。自动化平台,如Auto-GS通过迭代基因组筛选,将育种周期缩短了50%,在3到4年内成功培育出耐旱品种。此外,群体结构分层(例如3K-RG项目的9个亚群划分、中国6044份种质的5大生态区解析)进一步指导了种质资源的精准利用。
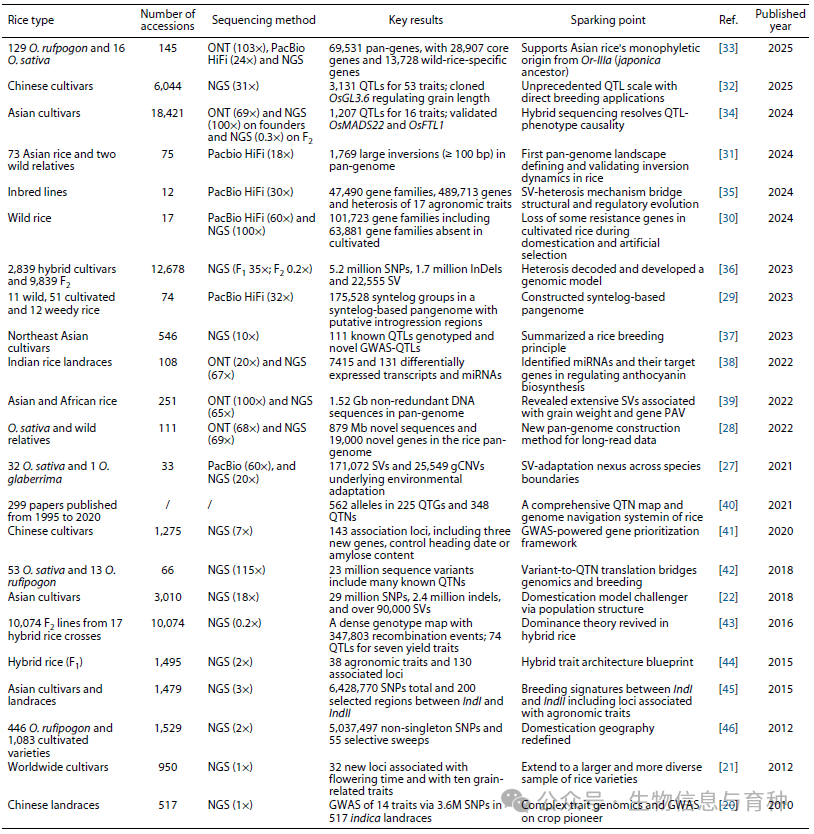
标志性水稻群体基因组学研究的总结。
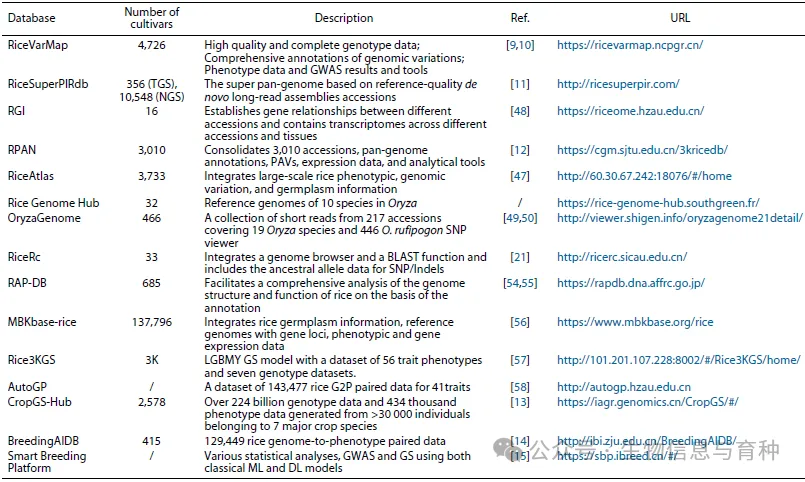
用于水稻群体基因组学数据存储及在线可视化功能的数据库。
目前,水稻群体基因组学研究仍面临大数据管理和模型可解释性的双重挑战。大规模种质测序产生了PB级数据(例如,5164份材料产生了60.78 Tb的数据),迫切需要云端架构和自动化管理来优化存储和计算流程。深度学习的“黑箱”问题阻碍了对生物学机制的解析,尽管新兴的植物专用大语言模型(LLM),如AgroNT(拥有10亿参数),在预测顺式调控元件方面取得了突破,但仍需解决可解释性危机。展望未来,水稻群体基因组学的突破点将在于三维基因组学与多模态AI的融合:染色质空间互作图谱将有助于解析非编码SVs的调控功能,从而支撑“结构智能育种”;跨尺度数据整合(基因组-转录组-表观组)将推动作物改良从“经验筛选”迈向“预测设计”,系统性挖掘野生种质的适应性模块(如野生稻LTR转座子),加速抗逆高产水稻的智能化开发。
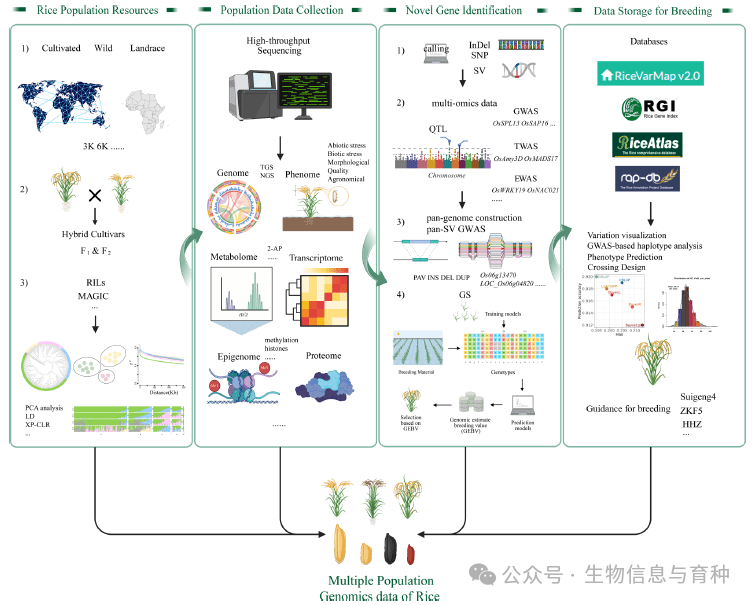
群体基因组学和多组学方法的整合框架促进水稻育种
浙江大学农业与生物技术学院博士研究生周宇涵与硕士研究生周子奇为本文的共同第一作者。
原文链接:
https://doi.org/10.48130/gcomm-0025-0012
